stacks简介、安装与使用
stacks简介、安装与使用
# stacks 简介
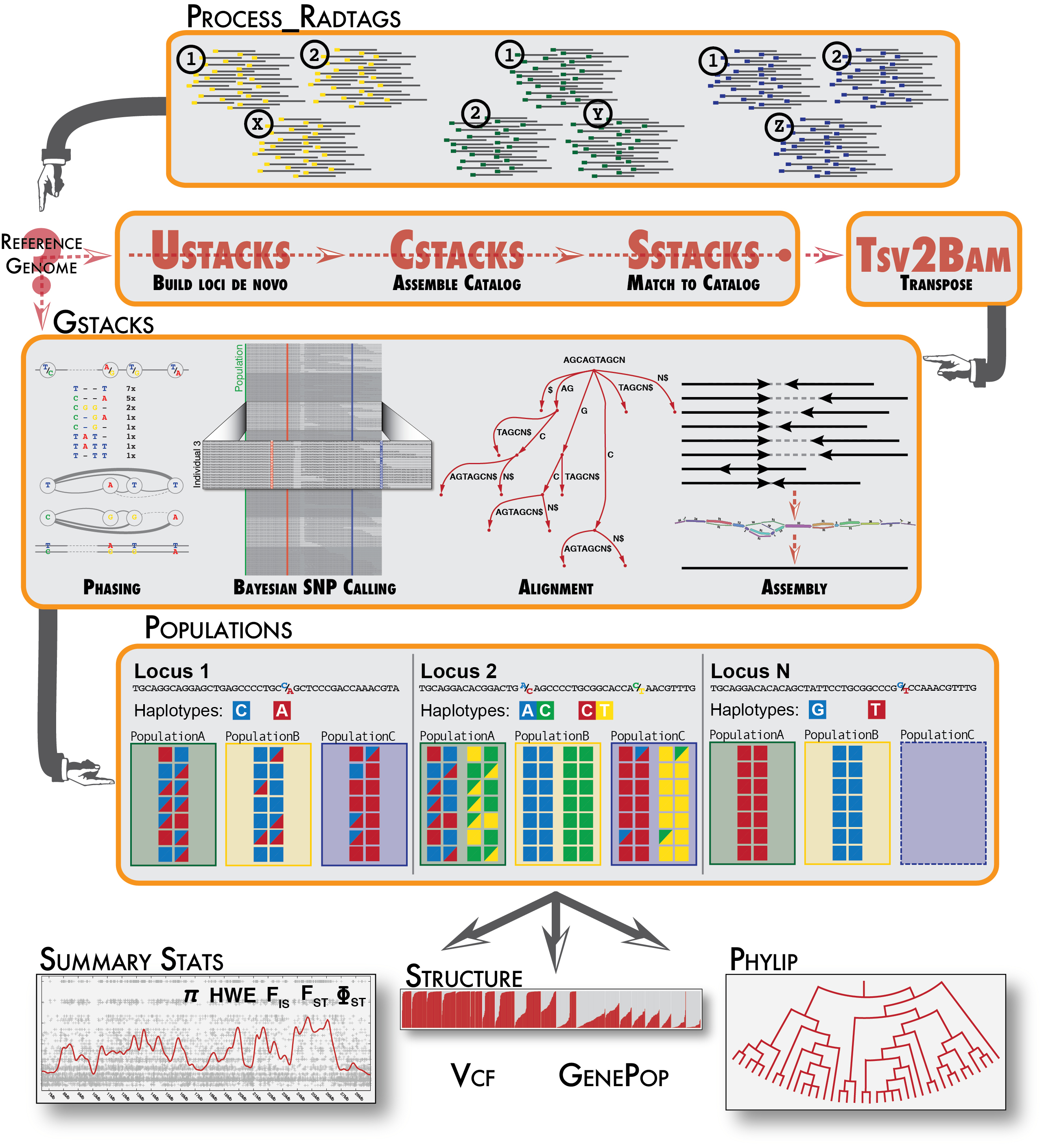
stacks被设计用于处理任何基于限制性酶的数据,如GBS,CRoPS,sdRAD和ddRAD。stacks被设计成一个模块化流程,有效地管理和组装来自多个样本的大量短读序列。stacks可以通过以下两种方式识别一组个体中的基因位点:无参de novo或者有参比对(允许gap),然后对每个基因位点进行基因型划分。stacks通过最大似然统计模型来识别序列多态性,并将其与测序错误区分开来。stack使用Catalog来记录一个群体中所有被识别的基因座,并将个体与Catalog相匹配,以确定在每个个体的每个基因座上存在哪些单倍型等位基因。 stacks是用c++实现的,使用Perl包装。基于OpenMP库实现其核心算法是多线程的,该软件可以处理来自数百个人的数据,包括数百万个基因型。
- stacks的de novo分析z分为六个主要阶段。第一,读取数据由process_radtags程序进行去重和清理。接下来的三个阶段包含了stacks最重要的流程:构建loci (ustacks)、创建loci的Catalog(cstacks)以及根据Catalog进行匹配(sstacks)。第五阶段,执行gstacks程序,对双端contigs进行组装,调取群体中的变异位点和每个样本的基因型。在最后阶段,执行种群程序,该程序可以过滤数据,计算种群遗传学统计数据,并导出各种数据格式。
- stacks中的有参分析分为三个主要阶段。process_radtags程序与de novo流程类似,然后执行gstacks程序,它将读取比对好的reads来组装基因座(并像在de novo那样对它们进行基因分型),然后执行种群程序。
# stacks 安装 (centos7 为例)
## stacks 需要gcc4.9+,centos7默认版本为4.8.5,建议使用scl工具安装高版本gcc环境
sudo yum install centos-release-scl scl-utils-build
sudo yum install devtoolset-9
scl enable devtoolset-9 bash
## 成功激活gcc9环境
wget http://catchenlab.life.illinois.edu/stacks/source/stacks-2.54.tar.gz
tar -zxf stacks-2.54.tar.gz
cd stacks-2.54/
## prefix选择合适的安装路径
./configure --prefix=/usr
make -j4
sudo make install
2
3
4
5
6
7
8
9
10
11
12
# stacks 使用(无参rad-seq)
# 一步完成
无参时,Stacks2可以用denovo_map.pl流程完成所有的分析内容。
输入:
PE fastq.gz文件,map_file.pop文件。
map_file.pop文件格式为:样本名[TAB]组名。如果没有群体分组信息,则所有样本的组名写成相同的名字
如果输入的是gz压缩的PE reads具体代码如下:
denovo_map.pl --samples 01.fastq/ --popmap map_file.pop -o stack_output/ -T 4 --paired
其中所有reads均存放在01.fastq目录下,并且必须命令为ID.1.fq.gz和ID.2.fq.gz,ID为样本id,例如sample1.1.fq.gz和sample2.2.fq.gz。并且fastq头行必须要有/1标识Reads 1,/2标识Reads 2,如下Reads1和Reads2中的头行
# 分步处理
# 前处理
如果fastq头行没有/1和/2标识,可采用Stack2自带的process_radtags完成前处理。当然,这个程序能做的事远不止转个格式,它还可以去除低质量的序列,demuliplexing,去除barcode等。stacks程序要求所有reads的长度必须一致,因此,此处对reads进行了长度筛选并裁剪到了相同的长度135bp。
mkdir 01_quality_control
mkdir 04_stacks_logs
for i in 00_rawdata/*.1.fastq.gz; do
echo '[ processing '${i}' ]' >> 04_stacks_logs/process_radtags.log
outdir=${i##*/}
outdir=${outdir/.clean*/}
mkdir 01_quality_control/${outdir}
echo ${outdir}
process_radtags -1 $i -2 ${i/.1.fastq/.2.fastq} -q -c -i gzfastq -t 135 --len-limit 135 -b barcode \
--disable-rad-check -o 01_quality_control/${outdir} >> 04_stacks_logs/process_radtags.log 2>&1
mv -T 01_quality_control/${outdir}/sample.1.fq.gz 01_quality_control/${outdir}.1.fq.gz
mv -T 01_quality_control/${outdir}/sample.2.fq.gz 01_quality_control/${outdir}.2.fq.gz
done
# 统计过滤后的reads数
mkdir 03_stats
for i in 01_quality_control/*gz; do
echo -en ${i##*/}'\t' >> 03_stats/read_number.tsv ; zcat $i |wc -l >> 03_stats/read_number.tsv
done
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
完成后会在Sample1目录下生成以下文件
process_radtags.00.RawData.log : 这个文件会记录下总的有多少条reads,因什么原因去掉reads,去掉了多少等信息
sample1.1.fq.gz和sample1.2.fq.gz : 这两个文件是过滤后可用于后续分析的文件
为了完成reads的组装(聚类),stacks采用两步法:
(1) 用ustacks分别对单个样本的R1进行组装(聚类),获取consensus序列。这一步生成的结果称为loci;
(2) 再使用cstacks将所有样本的loci聚合到一起。这一步生成的结果称为catalog。
在ustacks中控制组装结果的最关键参数是M,它表示在一个杂合样本中两个等位基因容许的mismatch数,默认为2;在cstacks中控制组装结果的最关键参数是n,它表示群体间等位基因容许的mismatch数,默认为1。这两个参数设置时差异不要过大,建议设置为相同的值.
# ustacks
mkdir 02_stacks_output
id=1
for i in 01_quality_control/*.1.fq.gz; do
echo $id
echo "[ processing "${i##*/}" ]" >> 04_stacks_logs/ustacks.log
name=${i##*/}
name=${name/.1.fq.gz/}
ustacks -t gzfastq -f ${i} -o 02_stacks_output -i ${id} --name $name\
-p 8 -M 4 >> 04_stacks_logs/ustacks.log 2>&1
let 'id+=1'
done
for i in 02_stacks_output/*tags.tsv.gz; do
echo -en ${i##*/}'\t' >> 03_stats/individual_stacks_number.tsv
zcat $i |grep consensus |wc -l >> 03_stats/individual_stacks_number.tsv
done
2
3
4
5
6
7
8
9
10
11
12
13
14
15
注意:在这里有个-i选项,它是整套分析流程中样本的ID,需为整数类型。
采用最大似然模型对每个样本单独组装,顺带给SNP calling。这步运行完成后每个样本生成三个文件,以sample1为例:sample1.tags.tsv.gz,sample1.snps.tsv.gz和sample1.alleles.tsv.gz。另外,在log文件中会记录下每个样本的深度情况。
tags.tsv用于存入assembled loci序列信息,各列解释:
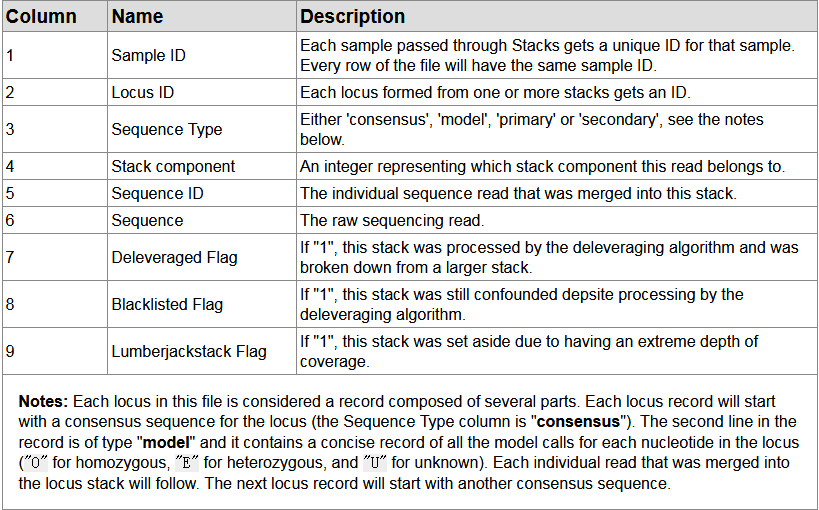 snps.tsv: Model calls from each locus,各列解释:
snps.tsv: Model calls from each locus,各列解释:
 alleles.tsv: Haplotypes/alleles recorded from each locus,各列解释:
alleles.tsv: Haplotypes/alleles recorded from each locus,各列解释:

# cstacks
cstacks -P 02_stacks_output -M map.pop -n 4 -p 8 >> 04_stacks_logs/cstacks.log 2>&1
cstacks采用kmer的办法来进行比对。ustacks生成的三个文件在这一步都会用到。这步运行完所有样本合在一起生成三个文件:catalog.allels.tsv.gz, catalog.snps.tsv.gz和catalog.tags.tsv.gz。其中最重要的是catalog.tags.tsv.gz,它是所有loci合在一起组装成的catalog,相当一个超级consensus文件。
ustacks的tags.tsv.gz文件中除记录有consensus序列外还有model和primary,但是cstacks的tags.tsv.gz文件中仅有consensus序列。其各列解释与ustacks文件中生成的对应文件相同。
# sstacks
sstacks -P 02_stacks_output -M map.pop -p 8 >> 04_stacks_logs/sstacks.log 2>&1
for i in 02_stacks_output/*.matches.tsv.gz; do
temp=${i##*/}
echo $temp
gunzip -c $i > 03_stats/${temp/.gz/}
done
cd 03_stats
# number of catalogs matched in each sample
for i in *.matches.tsv; do
echo -en ${i/.matches.tsv/}'\t' >> catalog_stats.tsv ; cat $i| cut -f 1 | uniq|wc -l >> catalog_stats.tsv
done
# number of locus matched in each sample
for i in *.matches.tsv; do
echo -en ${i/.matches.tsv/}'\t' >> loci_stats.tsv ; cat $i| wc -l>> loci_stats.tsv
done
cd ..
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
上一步cstacks生成的三个文件,这步均会用到。sstack将各样本的PEreads中R1序列match到上步cstacks生成的catalog上,运行完生成每个样本的matches.tsv.gz文件
matches.tsv: Matches to the catalog,各列解释:
 注:到这一步为止,以上各步,仅采用R1 reads。R2 reads在下一步才会用到
注:到这一步为止,以上各步,仅采用R1 reads。R2 reads在下一步才会用到
# tsv2bam
tsv2bam -P ./stack_output/ -M map_file.pop -t 4 -R /lustre/Work/project/resequencing/20180514_17141_xun/09.stacks/91.fastq
tsv2bam将sstacks生成的各样本tsv文件转为各locus的bam文件。运行完后,每个样本生成一个matchs.bam文件。其中-R参数用于引入双端reads但并未使用。
# gstacks
gstacks -P stack_output/ -M map_file.pop -t 4
gstacks主要用于merge PE reads成contig,然后将reads比对到locus上,并在群体水平call 变异以及分型。这步运行完生成catalog.fa.gz和catalog.calls,其中catalog.calls是个二进制文件。
# populations
populations -P stack_output/ -M map_file.pop --threads10 --fasta_samples --fasta_loci --vcf -O popdir/
populations程序在群体水平进行汇总统计(如计算Fst等),运行完生成以下文件:
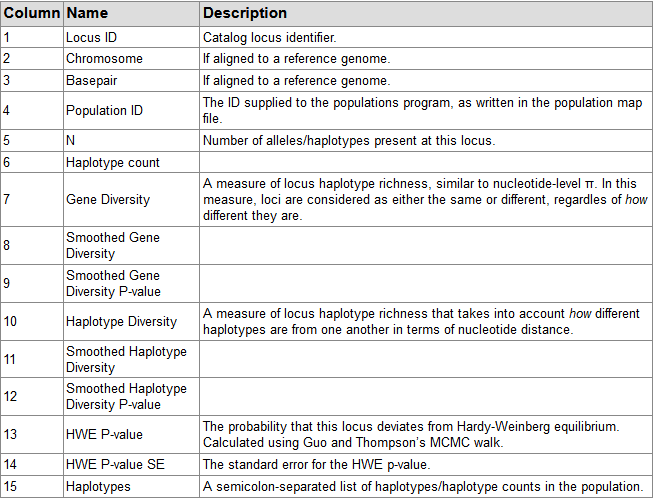
populations.sumstats.tsv: Summary statistics for each population,各列解释:
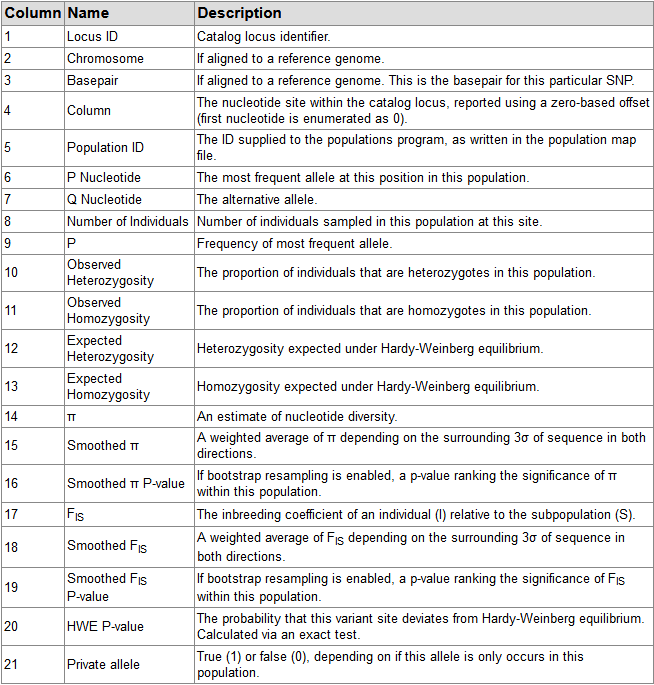
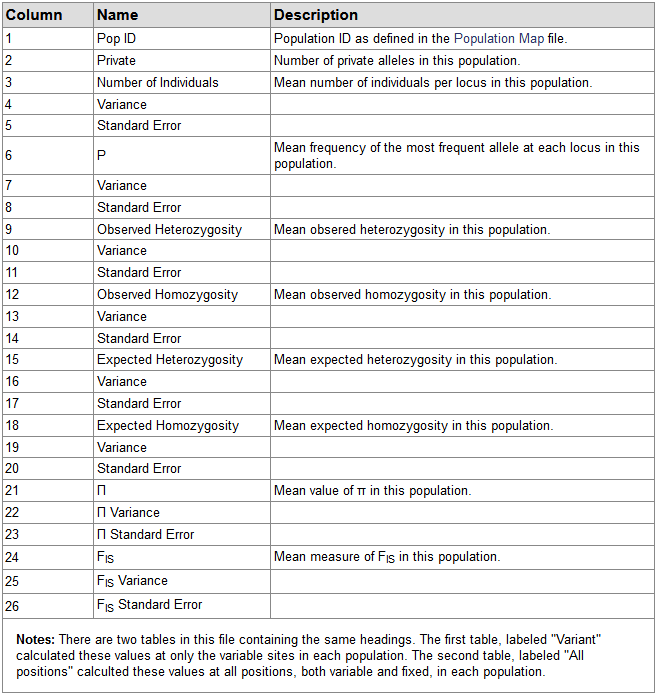
populations.sumstats_summary.tsv: Summary of summary statistics for each population,各列解释:
populations.samples.fa: Per-locus, per-haplotype sequences
如果指定--fasta_samples,才会生成该文件,该文件提供了全部RAD locus序列,这个文件和populations.haplotypes.tsv相对应。
populations.hapstats.tsv: Haplotype-based summary statistics for each locus in each population